ARTICULO ORIGINAL
REVISTA DE LA FACULTAD DE MEDICINA HUMANA 2020 - Universidad Ricardo PalmaDOI 10.25176/RFMH.v20i3.3040
CARACTERIZACIÓN CLÍNICOPATOLÓGICA, GENOTIPIFICACIÓN VIRAL Y HETEROGENEIDAD GENÉTICA COMO DETERMINANTES DE RIESGO EN COVID-19: DISEÑO DEL ESTUDIO Y HALLAZGOS INICIALES
CLINICAL-PATHOLOGICAL CHARACTERIZATION, VIRAL GENOTIPIFICATION AND GENETIC HETEROGENEITY AS RISK DETERMINANTS IN COVID-19: STUDY DESIGN AND INITIAL FINDINGS
Mauricio Postigo Mac Dowall1, Alejandro Barrionuevo-Poquet1, Oscar Carnero-Fuentes1
, Guido Pareja-Begazo1
, Claudio Coayla-Cano1, Aly Gallo-Lopez 2, Jhony A. De La Cruz-Vargas2
1Hospital Base Carlos Alberto Seguín Escobedo-EsSalud, Arequipa-Perú.
2Instituto de Investigación en Ciencias Biomédicas, Universidad Ricardo Palma, Lima-Perú.
RESUMEN
Introducción: El objetivo del estudio es describir las características clínicas, patológicas, virológicas y genéticas de la respuesta inmune de los pacientes diagnosticados con infección por SARS-CoV-2 y su relación con el curso desfavorable
de la enfermedad. Métodos: Estudio descriptivo, relacional, longitudinal y retrospectivo basado en la revisión de historias clínicas, toma de biopsias tru-cut post-mortem de pulmón e hígado, toma de muestras de sangre e hisopado naso-orofaríngeo
o de aspirado del tubo endotraqueal. En la primera fase las biopsias serán procesadas y estudiadas con histología convencional e inmunohistoquímica en el servicio de Anatomía Patológica del hospital Nacional Carlos Seguín Escobedo de Arequipa,
Perú. Resultados: La edad media avanzada, el sexo masculino y la presencia de comorbilidades fue predominante en los pacientes fallecidos. Las biopsias pulmonares mostraron predominantemente las fases exudativa y parcialmente proliferativa
del daño alveolar difuso y focal, y neumonía organizativa y fibrinoide aguda, asociada principalmente a una hiperplasia de macrófagos intraalveolares con acumulación dentro del espacio alveolar, semejando una neumonía descamativa, así como
neumocitos intraalveolares binucleados y atípicos, con nucléolos eosinofílicos (semejante a virocitos) en algunos casos. En la gran mayoría de casos se observaron depósitos de fibrina intravascular asociada al acúmulo de células inflamatorias
compuestas por neutrófilos y monocitos, representando microtrombosis. Las biopsias de hígado mostraron esteatosis predominantemente macrovesicular y en dos casos se observó esteatosis microvesicular. Adicionalmente, se observaron diversos
grados de necrosis e inflamación portal y lobular leve. Conclusión: Los hallazgos clínicos y patológicos en este primer reporte son consistentes con publicaciones previas y confirman el patrón de daño alveolar difuso asociado a agregados
de macrofagos intraalveolares y microtrombosis; ademas esteatosis macro y microvesicular hepatocitica, junto a grados variables de necrosis.
Palabras clave: SARS-COV-2; COVID-19; Patología; Inmunohistoquímica (fuente: DeCS BIREME).
ABSTRACT
Introduction: The objective of the study is to describe the clinical, pathological, virological and genetic characteristics of the immune response of patients diagnosed with SARS-CoV-2 infection and its relationship with the unfavorable
course of the disease. Methods: Descriptive, relational, longitudinal and retrospective study based on the review of medical records, taking post-mortem tru-cut biopsies of the lung and liver, taking blood samples and naso-oropharyngeal
swab or endotracheal tube aspirate. In the first phase, the biopsies will be processed and studied with conventional and immunohistochemical histology in the Pathological Anatomy service of the Carlos Seguín Escobedo National Hospital in Arequipa,
Peru. Results: Advanced mean age, male sex, and the presence of comorbidities were predominant in deceased patients. Lung biopsies showed predominantly the exudative and partially proliferative phases of diffuse and focal alveolar
damage, and acute organizational and fibrinoid pneumonia, associated primarily with intraalveolar macrophage hyperplasia with accumulation within the alveolar space-resembling desquamative pneumonia, as well as binucleated intraalveolar pneumocytes
and atypical, with eosinophilic nucleoli (similar to virocytes) in some cases. In the vast majority of cases, intravascular fibrin deposits associated with the accumulation of inflammatory cells composed of neutrophils and monocytes, representing
microthrombosis, were observed. Liver biopsies showed predominantly macrovesicular steatosis and in two cases microvesicular steatosis was observed. Additionally, varying degrees of necrosis and mild portal and lobular inflammation were observed.
Conclusion: The clinical and pathological findings in this first report are consistent with previous publications and confirm the pattern of diffuse alveolar damage associated with aggregates of intraalveolar macrophages and microthrombosis;
in addition, macro and microvesicular hepatocytic steatosis, together with variable degrees of necrosis.
Key words: SARS-COV-2; COVID-19; Pathology, Immunohistochemistry (source: MeSH NLM).
La pandemia ocasionada por la infección por el nuevo coronavirus 2019, o como también es conocido coronavirus del síndrome de falla respiratoria aguda severa 2019 (SARS-CoV-2019), está causando severos daños a los sistemas sanitarios y económicos a nivel mundial, con más de seis millones de infectados y más de trescientos ochenta mil fallecidos globalmente a la fecha(1). Esta enfermedad apareció hacia finales del año 2019 en Wuhan, una ciudad comercial en China central, en la que causó la muerte de más de mil ochocientas personas e infectó más de siete mil individuos en los primeros quince días de la epidemia. El comité internacional de la taxonomía de virus nombró el virus como SARS-CoV-2 y la enfermedad COVID-19.(1,2)
La familia de coronavirus (CoV) es una clase de virus RNA de cadena simple y encapsulado con un muy extenso rango de fuentes naturales. El SARS-CoV-2 es un miembro de los beta-coronavirus junto al SARS- CoV y MERS-CoV, con quienes comparte similitudes en su genoma, biología y clínica. Estos virus causan enfermedades respiratorias, entéricas, hepáticas y neurológicas. Hasta ahora han sido evaluados más de treinta y siete mil genomas del SARS-CoV-2 a nivel mundial según GISAID, encontrándose una gran cantidad de variantes genómicas.(3)
Se ha identificado que el sexo masculino, edad mayor de 65 años y el tabaquismo son factores de riesgo para la progresión de la enfermedad en pacientes con COVID-19. Las enfermedades subyacentes como hipertensión, diabetes, enfermedad cardiovascular y respiratoria son mayores en una proporción estadísticamente significativa en pacientes críticos y mortales comparados con los pacientes no críticos.(4)
Múltiples estudios demuestran que una inadecuada respuesta inflamatoria sistémica, predominantemente pro-inflamatoria, con la denominada tormenta de citoquinas, juega un rol muy importante en la patogénesis de la enfermedad, especialmente en la enfermedad crítica y muerte, relacionados con el síndrome de distrés respiratorio agudo y sepsis viral con falla multiorgánica.(5) La tormenta de citoquinas puede iniciar la sepsis viral y la injuria pulmonar inducida por la inflamación que lleva a otras complicaciones que incluyen neumonitis, síndrome de distrés respiratorio agudo (ARDS), falla respiratoria, shock, falla multiorgánica y muerte.(6)
El ARDS es una patología pulmonar que dificulta la entrada del oxígeno a la circulación, y se ha demostrado histopatológicamente ser la causa de la mortalidad de la mayoría de trastornos respiratorios, incluyendo los casos fatales de SARS-CoV, MERS- CoV y SARS-CoV-2. Se han encontrado más de 40 genes candidatos asociados al desarrollo de ARDS que incluyen ACE2, IL-10, factor de necrosis tumoral (TNF), y factor de crecimiento del endotelio vascular.(7) Esto podría explicar la enfermedad severa y muerte de pacientes sin aparentes factores de riesgo de pronóstico negativo y la respuesta inmune inadecuada asociada a la progresión de la enfermedad.
Existen varias publicaciones que describen las características clínicas y epidemiológicas; sin embargo, los reportes de las características histológicas, así como la demostración de la presencia del virus en el tejido son relativamente limitados. Los reportes que describen las características histopatológicas evidencian en el pulmón cambios correspondientes principalmente a daño alveolar difuso y microtrombosis, necrosis focal de miocitos cardiacos, cambios degenerativos inespecíficos en otros órganos, siendo semejantes a lo observado en la infección por los virus SARS-CoV y MERS-CoV.(8-12) La patología es una herramienta que pone en evidencia los mecanismos fisiopatológicos interactuantes en los tejidos afectados por una enfermedad.
En este trabajo vamos a tratar de dilucidar las características del huésped y del agente infeccioso que podrían tener una relación con el pronóstico de los pacientes infectados con SARS-CoV-2, que incluye el describir las características clínicas y laboratoriales relacionadas al pronóstico ya definidas, la presencia de polimorfismos en genes inflamatorios y epiteliales que tienen una relación con la susceptibilidad a desarrollar ARDS, genotipificar mediante secuenciamiento de próxima generación los SARS-CoV-2 a partir de hisopado nasofaríngeo o aspiración de tubo endotraqueal, el estudio histopatológico de las biopsias de pulmón, hígado y posiblemente riñón u otros órganos así como identificar la presencia del SARS-CoV-2 mediante RT-PCR en estos tejidos a partir del tejido embebido en parafina. Se buscará definir una relación entre estos factores y el curso de la enfermedad dividida entre enfermedad leve (ambulatorio), en pacientes hospitalizados (enfermedad moderada a grave), en cuidados intensivos (enfermedad crítica) y pacientes fallecidos.
Creemos que la comprensión de los mecanismos patogénicos es indispensable para un tratamiento racional y eficaz de las enfermedades. Dado que se espera que una gran parte de la población se infecte con el SARS-CoV-2, consideramos que es muy importante determinar qué factor o factores y en qué tipo de pacientes la enfermedad va a tener una progresión negativa, y va ocasionar un daño severo a la sociedad, al sistema sanitario y a la economía, para tomar medidas apropiadas dirigidas a la prevención primaria, secundaria y terciaria, incluyendo el uso de medicamentos adecuados para un tratamiento racional.
Para esto se aplicarán estrictos protocolos de bioseguridad de toma de biopsias y manejo de muestras, así como el consentimiento informado para la realización de este procedimiento y la toma de biopsias.
El objetivo general del estudio es evaluar las características clínicas, patológicas, virológicas y genéticas de la respuesta inmune de los pacientes diagnosticados con infección por SARS-CoV-2 y buscar una relación de estas con el curso desfavorable de la enfermedad.
MÉTODOS
Fases del proyecto
Hemos dividido el trabajo en tres fases. En la primera fase, nuestros objetivos consistirán en evaluar las características clinicoepidemiológicas de los pacientes infectados por SARS-CoV-2 con todos los grados de severidad de la enfermedad, las características histopatológicas de las biopsias tru-cut de pulmón, hígado y otros órganos de pacientes fallecidos con infección por SARS-CoV-2 y determinar la presencia de SARS-CoV-2 en las muestras de tejido por biopsia tru-cut mediante RT-PCR.
En la segunda fase buscaremos determinar el genotipo del SARS-CoV-2 en muestras de hisopado nasofaríngeo y/o aspiración del tubo endotraqueal mediante secuenciación de próxima generación en los pacientes infectados por SARS-CoV-2 en los diversos grados de severidad de la enfermedad.
En la tercera fase buscaremos determinar la presencia de polimorfismos u otras alteraciones genómicas en los genes relacionados a la respuesta inflamatoria y riesgo de distrés respiratorio en los pacientes infectados por SARS-CoV-2 de los diversos grados de severidad de enfermedad.
En las fases dos y tres se buscara una relación entre el genotipo del virus y de los genes de la respuesta inflamatoria y riesgo de ARDS con la severidad de la enfermedad.
Tipo y diseño general del estudio
El estudio es un trabajo descriptivo correlacional, longitudinal y retrospectivo basado en la revisión de historias clínicas, toma de muestras (sangre e hisopado naso-orofaríngeo o de aspirado del tubo endotraqueal) y el estudio de láminas con secciones histológicas e inmunohistoquímica en el servicio de Anatomía Patológica del hospital Nacional Carlos Seguín Escobedo (HBCASE) a partir de biopsias tru- cut de pulmón, hígado y otros órganos a los pacientes fallecidos en la unidad de cuidados intensivos (UCI) y hospitalización de medicina interna.
Universo de estudio, selección y tamaño de muestra
La población estará constituida por los casos diagnosticados de infección por SARS-Cov-2 mediante el método molecular (RT-PCR) o prueba rápida en la red asistencial Arequipa de EsSalud, con enfermedad leve/moderada, severa, crítica y fallecidos. No se realizará muestreo, se trabajará con la población por ser esta pequeña y accesible. Las historias clínicas se revisarán mediante su extracción del área de historias clínicas y de hospitalización o UCI de la red asistencial Arequipa, así como del sistema de gestión. Se evaluarán las láminas de histología en el Servicio de Anatomía Patológica del HBCASE. Las pruebas moleculares en sangre e hisopado nasofaríngeo se realizarán en cuarenta pacientes, las cuales se remitirán a un laboratorio de biología molecular externo que cuente con secuenciamiento genético.
Protocolo del estudio
Estudio histopatológico de las biopsias:
La primera fase del estudio se centrará en el estudio de los pacientes con COVID-19 en los que se realice biopsias post-mortem en la UCI. Se describirán las características clínicas y laboratoriales mediante la revisión de las historias clínicas. Las características laboratoriales se extraerán de las historias clínicas o del sistema de gestión. La toma de biopsias por aguja tru- cut se realizarán en la UCI inmediatamente posterior al fallecimiento del paciente. Las muestras se remitirán al servicio de Anatomía Patológica del HBCASE donde se les realizará el procesamiento histológico convencional y tinciones de inmunohistoquímica para definir las poblaciones celulares inflamatorias, que incluyen panqueratina, CD3, CD20, CD4, CD8, granzima B, mieloperoxidasa (MPO), CD61, CD56, CD138, CD117 y CD68.
Identificación del virus en las biopias tisulares:
En esta primera fase se realizará, adicionalmente, el estudio mediante RT-PCR para la identificación del genoma viral en los tejidos biopsiados. Para la realización de la RT-PCR del tejido se inicia el procedimiento con la calificación de la muestra por histopatología para definir la presencia de parénquima tisular con cambios patológicos. Se realizarán 5 cortes de 10µm, de los que se realizará la extracción del RNA viral, o en su defecto mediante previa evaluación en fresco del tejido mediante microscopía de luz. Se prepara el mix con kits definidos que cubran una región de screening y una definitiva. Luego se realizará la amplificación del protocolo del termociclador en tiempo real y análisis de resultados.
Secuenciamiento de próxima generación para la genotipificación viral y de la respuesta inflamatoria/ riesgo de distrés respiratorio agudo:
La segunda fase comprenderá el estudio mediante secuenciamiento de próxima generación de los hisopados de nasofaringe y orofaringe o aspiración del tubo endotraqueal de los pacientes positivos para infección por SARS-CoV-2. La tercera fase comprenderá el estudio mediante secuenciamiento de próxima generación de muestras de sangre periférica de los pacientes positivos para infección por SARS-CoV-2 en aproximadamente 40 pacientes para la determinación de polimorfismos de genes de la respuesta inflamatoria y de predisposición al desarrollo de síndrome de distrés respiratorio agudo y sepsis.
Se extraerá una muestra de 2ml de sangre periférica en tubos PAXgene® (BD Biosciences), 2 ml de tubo heparina y un hisopado nasofaringeo. Se realizará una segunda colección de sangre a los cinco días de hospitalización. Se extraerá DNA de las muestras de sangre periférica colectadas en los tubos PAXgene® usando el kit AllPrep DNA/RNA Mini kit (Qiagen). Las muestras de sangre periférica obtenidas en tubos de heparina serán centrifugadas para la obtención de plasma y preservadas a -80. Se usará el kit Magmax Viral Isolation (Thermofisher) para la extracción de ARN de SARS-CoV-2 (hisopado). Se evaluará la calidad y concentración del ARN. Se convertirá el ARN extraído en cDNA utilizando el kit SuperScript™ VILO™ cDNA Synthesis. Se procederá a preparar manualmente las
librerías genéticas a partir del cDNA de sangre y del virus. Para las librerías de sangre periférica se utilizará el panel de inflamación Ion AmpliSeq RNA Inflammation Respone Research (683 genes de respuesta inmune). Para las librerías provenientes de las diferentes cepas de SARS-CoV-2 se utilizará el panel Ion AmpliSeq SARS-CoV-2 que contiene 237 amplicones específicos para SARS-CoV-2 (99% del genoma). La preparación del templado y carga del chip Ion Chip 540 se realizará en el equipo Ion Chef. Las corridas de secuenciación se realizarán en el equipo Ion GeneStudio S5. El análisis de la data primaria se realizará el software Torrent Suite. Con la matríz de expresión se determinará los genes diferencialmente expresados y el análisis de enriquecimiento de genes entre pacientes con evolución favorable y aquellos que desarrollaron COVID-19 severo. Se correlacionará el perfil inmunológico de huésped con las variantes genéticas de SARS-CoV-2. Se utilizará un modelo estadístico predictivo para establecer un perfil inflamatorio/viral que determine la progresión a COVID-19 severo. Para los genes no incluidos en este kit (incluyendo ACE-2, HMOX2, ANGPT2, SOD3), se utilizará RT-PCR con la metodología antes mencionada.
Para las segunda y tercera fases se describirán las características clínicas y laboratoriales de los pacientes positivos para infección por SARS-CoV-2 mediante prueba molecular o prueba rápida tanto ambulatorios como hospitalizados mediante coordinación con las áreas de epidemiología, medicina interna y UCI. En los pacientes no hospitalizados se realizará interrogatorio directo, y en los hospitalizados y fallecidos se revisarán las historias clínicas. Las características laboratoriales se extraerán de las historias clínicas o del sistema de gestión.
Procedimientos para garantizar aspectos éticos
El presente trabajo beneficiará a la institución y a nuestros pacientes: busca determinar qué factores están relacionados a un desenlace negativo y fatal de los pacientes infectados por SARS-CoV-2, por lo que, de encontrarse una relación con el factor genético del huésped o del virus, podría buscarse la toma de medidas de prevención y terapia apropiadas. No existen publicaciones semejantes en la literatura médica actual, por lo que sería un aporte muy significativo al estudio y entendimiento de esta enfermedad a nivel global.
El presente estudio no tiene la finalidad de modificar el diagnóstico o tratamiento de los pacientes involucrados, así como el curso de la enfermedad. El presente trabajo involucra la participación de seres humanos y sus muestras biológicas. Se reclutarán aproximadamente un total de 40 casos para secuenciamiento genético y 40 casos para la toma de biopsias postmortem, sin rango de edades determinado. No se realizará ningún tipo de discriminación de pacientes.
Se utilizarán hojas de consentimiento informado para los pacientes vivos y para los familiares de pacientes que ingresen a hospitalización. El consentimiento informado para los pacientes a los que se vaya a realizar el estudio de secuenciamiento genético viral y del genoma inflamatorio, ya que se tomará a personas con enfermedad leve hasta crítica o fatal, se realizará directamente al paciente cuando sea posible y a los familiares cuando el paciente no sea capaz de otorgarlo (esté en condición crítica o fallezca). En caso el consentimiento sea obtenido de los familiares, luego del fallecimiento de los pacientes, se considerará el orden que establece el artículo 13 del código civil para decidir con respecto a una necropsia, incineración y sepultura; es decir, primero el cónyuge, luego descendientes, y luego ascendientes o hermanos.
No se realizará el pago a los participantes en el estudio. Se realizará un informe final a los pacientes o familiares de sus resultados en caso de que esto se autorice por los pacientes o sus familiares.
La realización de este trabajo no conllevará al daño de los involucrados, tanto personal de salud como a los pacientes, ya que se realizará bajo protocolos de manejo definidos, ni a terceros.
Los resultados de este estudio buscarán ser publicados en una revista médica indexada, así como será entregada a las autoridades respectivas de EsSalud según se solicite.
El presente trabajo de investigación mantendrá la confidencialidad de los nombres y otros medios de identificación personal de los pacientes dándoles un código propio del estudio. Se recogerá la identificación de los pacientes mediante nombres y apellidos, se les codificará inmediatamente y se les manejará de acuerdo a esta codificación. La lista con la identificación personal y su codificación será únicamente manejada por el investigador principal. Los investigadores secundarios únicamente manejarán la lista codificada, cumpliéndose con la ley de protección de datos personales.
Dado que las muestras para el estudio genético serán llevadas a un laboratorio externo, se establece que las muestras y la información genética no serán almacenadas ni se les extraerá información adicional a los fines concernientes al presente estudio.
El presente estudio cuenta con la aprobación del Comité de Ética en Investigación Específico para COVID-19 institucional.
RESULTADOS
Las características clínicas y patológicas de los casos iniciales se encuentran resumidas en la Tabla 1.
Estado situacional de COVID-19 en la red de Essalud de Arequipa
Hasta el momento (4 de Junio) en la región Arequipa, se han registrado 4 067 casos positivos y 85 muertes por esta enfermedad. En la red asistencial de EsSalud de Arequipa, el HBCASE se ha convertido en el hospital de referencia para COVID-19 en la seguridad social. En este hospital contamos con 158 camas de hospitalización y 23 camas UCI para pacientes con COVID-19. El número de pacientes hospitalizados entre los meses de abril – mayo 2020 fue de 136 y hasta el momento se han registrado 55 muertes por esta causa en el HBCASE. De los 136 casos hospitalizados, el 60% corresponde a varones y 40% a mujeres. En cuanto a la distribución por grupo etario, el 58% correspondió a personas por encima de los 60 años.
Descripción clínica de los primeros casos evaluados
Hasta el momento hemos incluido en nuestro estudio siete pacientes fallecidos en la UCI a los que hemos realizado las biopsias con aguja tru-cut. La mayoría fueron de sexo masculino (n: 4, 57%), el promedio de edad fue de 75 años, el 42% de pacientes tenía sobrepeso, 42%tenían al menos unacomorbilidad, que en orden de frecuencia fueron: hipertensión arterial (28%), enfermedad renal crónica (28%), y enfermedad pulmonar intersticial difusa (14%). En cuanto al cuadro clínico de ingreso, el tiempo de enfermedad antes del ingreso fue de 6,8 días en promedio, el síntoma más frecuente fue disnea; tos y sensación de alza térmica fueron los síntomas adicionales en frecuencia. En cuanto al hemograma, la mayoría presentaron en su evolución leucocitosis con neutrofilia y linfopenia. La relación neutrófilos: linfocitos fue marcadamente elevada. Los pacientes desarrollaron enfermedad severa e ingresaron a la UCI en promedio a los 8,1 días después del inicio de la enfermedad. En cuanto a las escalas de severidad, el promedio de score APACHE II de los pacientes fue de 13,4 puntos; los puntajes de SOFA al ingreso, tercer y séptimo día fueron de 6, 8 y 7 respectivamente. Todos los pacientes fueron sometidos a ventilación mecánica, el promedio de la relación entre pao2 a fio2 (pao2/fio2) al primer, tercer y sétimo día fue de 136, 171 y 190, respectivamente. La distribución de severidad del síndrome de distrés respiratorio agudo, usando la definición de Berlín, sin corregir para la altura, fue moderado en la mayoría de casos (85%). Las fallas orgánicas desarrolladas por los pacientes fueron, en orden de frecuencia: falla renal aguda en el 85%, falla hepática aguda en el 42% y falla cardiaca aguda en el 14% de los casos. Se obtuvieron dos cultivos de secreción bronquial positivos, uno con A. baumanii multidrogo resistente (MDR) y otro con P. aeruginosa MDR, y adicionalmente un hemocultivo positivo para A. baumanii en un tercer paciente. El promedio de días de estadía en la UCI de los pacientes fue de once días.
Hallazgos patológicos de los primeros pacientes
Las biopsias pulmonares mostraron predominantemente una hiperplasia de macrófagos intraalveolares con acumulación dentro del espacio alveolar, que fue variable, desde leve a severa; adicionalmente, se observa una leve a moderada hiperplasia de los neumocitos intraalveolares con células de tamaño grande y formas binucleadas y atípicas, con nucléolos eosinofílicos (semejante a virocitos) en algunos casos. Varios casos presentaron depósitos de material fibrinoide intraalveolar en agregados con formación de membranas hialinas, que fueron focales. Adicionalmente, se observa un leve a moderado infiltrado inflamatorio compuesto predominantemente de histiocitos/monocitos y neutrófilos en los septos interalveolares, confirmados con las tinciones inmunohistoquímicas para CD68, CD4 y mieloperoxidasa. Con respecto al infiltrado linfoide, en todos los casos fue muy leve compuesto por células T predominantemente CD8-positivas y en dos que mostraron una inflamación severa se evidenciaron linfocitos T CD4 y CD8. No fueron casi evidentes linfocitos B CD20-positivos, células plasmáticas CD138-positivas, células natural killer CD56-positivas, mastocitos CD117-positivas. En la gran mayoría de casos se observaron depósitos de fibrina intravascular asociada al acúmulo de células inflamatorias compuestas por neutrófilos y monocitos, representando microtrombosis.
Las biopsias de hígado mostraron esteatosis predominantemente macrovesicular y dos casos mostraron esteatosis microvesicular. Adicionalmente, se observaron diversos grados de necrosis e inflamación portal y lobular leve.
Tabla 1. Características clínicas y patológicas de los casos iniciales.
| N° | Edad | Sexo | IMC | Comorbilidades | Tiempo desde inicio de síntomas hasta el desarrollo de ARDS | Alteraciones hematológicas | Relación neutrófilos: linfocitos inicio y final | Serevidad ARDS | Falla aguda de órgano blanco | Score apache 2 | Score sofa | Tratamiento recibido (Días) | Principales hallazgos patológicos pulmonares |
Principales hallazgos patológicos hepáticos |
| 1 | 71 | M | Sobrepeso | No | 8 | Leucocitosis con neutrofilia y linfopenia | 17-6,8 | Severa | Renal y hepática | 15 | 11,5 | HCQ, L/R,C (3) | Membranas hialinas, hiperplasia y desprendimiento de neumocitos, hiperplasia de macrófagos alveolares, microtrombosis, células multinucleadas. | Esteatosis macrovesicularynecrosis hepatocíticafocal. |
| 2 | 83 | M | Normal | HTA, ERC | 7 | Leucocitosis con neutrofilia y linfopenia | 22-47 | Moderada | Renal, hepática y cardíaca | 14 | 8 | HCQ, L/R,C (2,2,3) | Membranas hialinas, hiperplasia y desprendimiento de neumocitos, microtrombosis, células multinucleadas, fibrina y hemorragia intraalveolar. | Esteatosis microvesicular,necrosis difusa. |
| 3 | 76 | M | Normal | No | 8 | Leucocitosis con neutrofilia y linfopenia | 30-32 | Moderada | Renal | 16 | 6,5 | L/R (4) | Hiperplasia y desprendimiento de neumocitos, hiperplasia de macrófagos alveolares, microtrombosis, células multinucleadas, fibrina y hemorragia intraalveolar. | Esteatosis microvesicular |
| 4 | 71 | M | Sobrepeso | ERC, EPID | 8 | Leucocitosis con neutrofilia y linfopenia | 10-12 | Moderada | Renal y hepática | 15 | 7,5 | No | Esteatosis microvesicular | |
| 5 | 79 | F | Sobrepeso | No | 6 | Anemia leve, leucositosis con neutrofilia y linfopenia | 6-11 | Moderada | Renal, hepática y cardíaca | 11 | 4,3 | HCQ, L/R,C (1,1,3) | Membranas hialinas, fibrina y hemorragia intraalveolar, hiperplasia y desprendimiento de neumocitos. | Esteatosis microvesicular |
| 6 | 67 | F | Normal | No | 14 | Anemia leve, leucositosis con neutrofilia y linfopenia | 12-Feb | Moderada | Renal | 12 | 8,3 | C (3) | Hiperplasia y desprendimiento de neumocitos, hiperplasia de macrófagos alveolares, microtrombosis, células multinucleadas, fibrina y hemorragia intraalveolar. | Esteatosis microvesicular |
| 7 | 80 | F | Normal | HTA | 4 | Linfopenia | 7,4-7,7 | Moderada | No | 11 | 6,6 | No | Hiperplasia y desprendimiento de neumocitos, hiperplasia de macrófagos alveolares, microtrombosis, células multinucleadas, fibrina y hemorragia intraalveolar |
HTA: Hipertensión arterial, ERC: Enfermedad renal crónica, EPID: Enfermedad pulmonar intersticial difusa, HCQ: Hidroxicloroquina, L/R: Lopinavir/ritonavir, C: Corticoides.
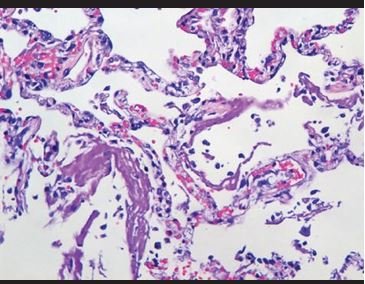 Figura 1. Depósitos de material hialino intraalveolar y congestión vascular formando membranas hialinas. |
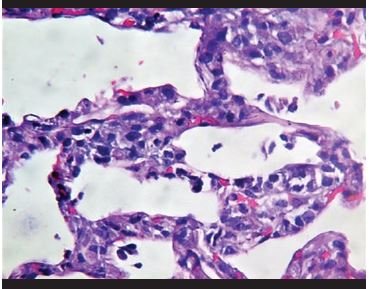 Figura 2. Infiltrado inflamatorio de neutrófilos e histiocitos en los septos interalveolares. |
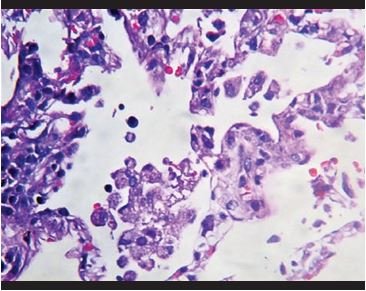 Figura 3. Hiperplasiademacrófagosintraalveolares. |
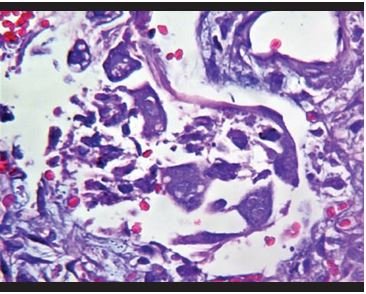 Figura 4. Células epiteliales binucleadas con nucléolos grandes eosinofílicos, semejantes a virocitos. |
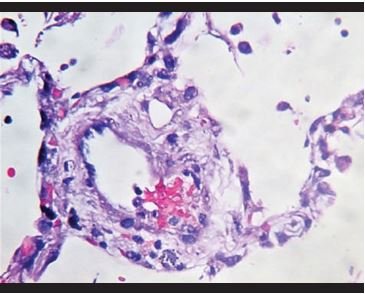 Figura 5. Congestión vascular con depósitos de fibrina y adherencia de células inflamatorias a la pared vascular. |
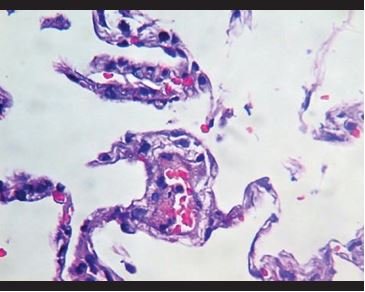 Figura 6. Congestión vascular con depósitos de fibrina y adherencia de células inflamatorias a la pared vascular. |
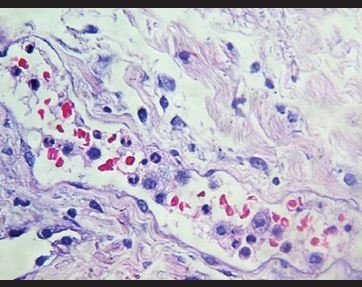 Figura 7. Congestión con neutrófilos y monocitos intravasculares. |
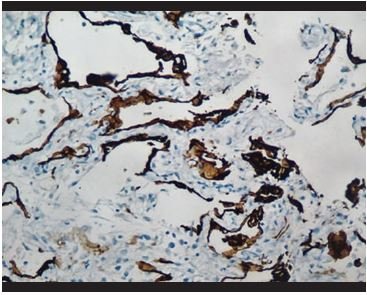 Figura 8. Tinción inmunohistoquímica para panqueratina (AE1/AE3). Se evidencia hiperplasia y desprendimiento de neumocitos alveolares. |
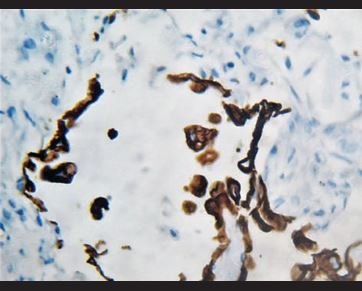 Figura 9. Tinción inmunohistoquímica para panqueratina en la que se evidencia que las células atípicas son positivas, correspondiendo a células de estirpe epitelial. |
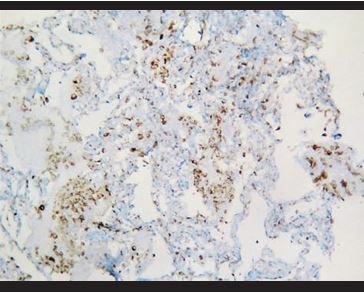 Figura 10. Tinción inmunohistoquímica para CD68, en la que se evidencia la presencia de agregados de macrófagos intralveolares. |
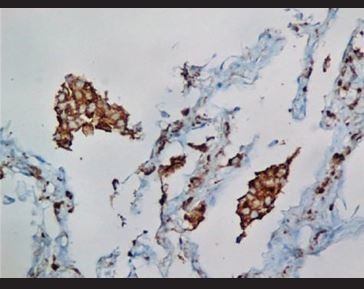 Figura 11. Tinción inmunohistoquímica para CD68, en la que se evidencia la presencia de agregados de macrófagos intralveolares en un patrón de neumonitis descamativa. |
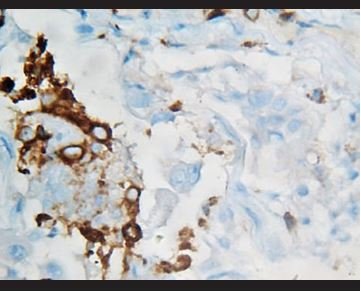 Figura 12. Tinción inmunohistoquímica para CD68, en la que se evidencian macrófagos intraalveolares positivos, mientras que las células binucleadas atípicas son negativas. |
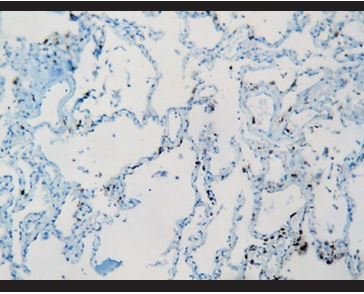 Figura 13. Tinción inmunohistoquímica para CD3: Escasos linfocitos T intersticiales. |
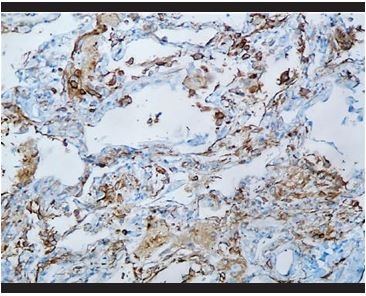 Figura 14. Tinción inmunohistoquímica para CD4: Presencia de hiperplasia de histiocitos intra- alveolares e intersticiales. |
 Figura 15. Tinción inmunohistoquímica para Mieloperoxidasa: Presencia de neutrófilos en el tejido intersticial peri-alveolar. |
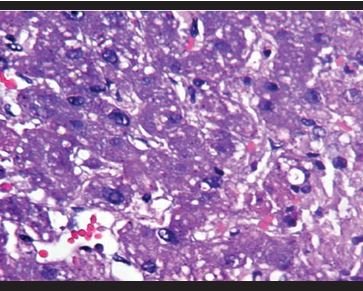 Figura 16. Hígado: Esteatosis microvesicular. |

Figura 17. Hígado: Esteatosis macrovesicular con degeneración balonizante hepatocítica y necrosis focal.
Los siete pacientes fallecidos iniciales fueron predominantemente de sexo masculino, edad media avanzada, mostraron sobrepeso, y comorbilidades como hipertensión arterial, enfermedad renal crónica y enfermedad pulmonar intersticial difusa. Adicionalmente, en el hemograma se evidenció en la mayoría leucocitosis con neutrofilia y linfopenia, con un elevado índice neutrófilos:linfocitos en todos los pacientes. Importantemente, todos presentaron al final falla renal aguda, y en menor grado cardíaca. Las biopsias pulmonares confirman el compromiso reportado en estudios previos correspondientes al patrón de daño alveolar difuso con injuria epitelial y endotelial, con el agregado que en nuestros casos fue muy prevalente la presencia de un importante
componente inflamatorio macrofágico con agregados intra-alveolares; también fue notoria la presencia de neutrófilos en las paredes alveolares y en el intersticio. El componente linfocitario fue mínimo y representado por células T CD4 y CD8 positivas. También fue evidente la presencia de microtrombosis en los vasos de pequeño calibre. Las biopsias hepáticas mostraron esteatosis macro y microvesicular, junto con grados variables de necrosis.
CONCLUSIÓN
Estos casos representan el primer paso del estudio.Los hallazgos clínicos y patológicos son consistentes con publicaciones previas y confirman el patron de daño alveolar difuso. Esperamos completarlo con un mayor número de casos y el resto del protocolo de biología molecular, lo que creemos aportará un conocimiento importante sobre la fisiopatología de esta enfermedad.
Contribuciones de autoría: Los autores participaron en la génesis de la idea, diseño de proyecto, recolección e interpretación de datos, análisis de resultados y preparación del manuscrito del presente trabajo de investigación.
Financiamiento: EL proceso de financiamiento de los estudios moleculares está en proceso.
Conflicto de interés: Los autores declaran no tener conflicto de interés.
Recibido: 03 de junio 2020
Aprobado: 10 de junio 2020
Correspondencia: Mauricio Postigo Mac Dowall
Dirección: Servicio de Anatomía Patológica, hospital Carlos Seguín Escobedo-EsSalud. Esq. Peral y FIltro s/n Arequipa, Arequipa-Perú.
Teléfono: 958348121
Correo electrónico: maupostigomd80@gmail.com
REFERENCIAS BIBLIOGRÁFICAS
